金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
关于深度学习在生物信息学中的应用这个话题由来已久,并已经有一些实际的案例。趁着DeepMind团队通过AlphaFold2算法预测蛋白质三维结构取得突破成就的契机,再来谈一谈这个话题。
可能你并不是很了解BP、CNN、RNN等这类算法的原理,但你依然对于深度学习在生物信息学应用感兴趣,阅读这篇文章是一个不错的选择。我会省略掉数学推导,以一种纯科普的文字来进行阐述。
先看一看AlphaFold2的思路做一个调味菜吧:AlphaFold2 解决了蛋白质结构问题吗?DeepMind 解决这项生物学五十年难题有何重大意义?
下面开始正题,先用一篇文章讲述一个生物中深度学习的美妙故事,在2018年,nature在线发表了长文《Deep learning for biology》
Deep learning for biology 一种流行的人工智能方法为生物数据的调查和分类提供了有力的工具。但对于外行来说,这项技术带来了巨大的困难。
大脑的神经网络长期以来一直激励着人工智能研究人员
四年前,来自谷歌的科学家们出现在神经学家Steve Finkbeiner的家门口,这些研究人员来自位于加州山景城的谷歌加速科学研究部门,该部门旨在利用谷歌技术加速科学发现。他们对将“深度学习”方法应用于Finkbeiner的团队产生的大量成像数据很感兴趣,Finkbeiner的团队位于旧金山的格莱斯顿神经疾病研究所,也位于加州。
深度学习算法从一个极其庞大的带注释的数据集(比如一组图像或基因组)中提取原始特征,并利用它们创建一个基于隐藏其中的模式的预测工具。一旦经过训练,算法就可以应用这些训练来分析其他数据,有时这些数据来自非常不同的来源。
Finkbeiner说,这项技术可以用来“解决非常困难、棘手、复杂的问题,并能够看到数据中的结构——对于人类大脑来说,这些数据的数量太大、太复杂,无法理解”
他和他的团队使用被称为机器人显微镜的高通量成像策略生成大量数据,这是他们为研究脑细胞而开发的。但该团队无法以获取数据的速度分析数据,因此芬克贝纳欢迎合作的机会。
他表示:“老实说,当时我不能明确地说,我对深度学习可能解决的问题有什么把握,但我知道,我们生成数据的速度是我们分析数据的速度的两到三倍。”
如今,这些努力开始有了回报。芬克贝纳的团队和谷歌的科学家们用两组细胞训练了一种深度算法,其中一组细胞被人为标记,以突出科学家们通常看不到的特征,另一组细胞则没有标记。芬克贝纳说,当他们后来将算法暴露在从未见过的未标记细胞的图像上时,“它在预测这些图像的标签方面表现得惊人地好。”一份详细介绍这项工作的出版物正在出版中。
Finkbeiner的成功凸显了深度学习这一最有前途的分支是多么的重要,人工智能(AI)的研究,正在侵入生物学。这些算法已经渗透到智能手机、智能扬声器和自动驾驶汽车的现代生活中。在生物学中,深度学习算法能以人类无法做到的方式深入数据,探测出原本不可能捕捉到的特征。研究人员正在使用算法对细胞图像进行分类,建立基因组连接,推进药物发现,甚至发现不同数据类型之间的联系,从基因组学和成像到电子医疗记录。
在bioRxiv上有超过440篇文章讨论深度学习;PubMed在2017年列出了700多条参考文献。生物学家和临床研究人员正在广泛使用这些工具。但研究人员在理解这些算法的作用以及确保它们不会把用户引入歧途方面面临着挑战。
训练智能算法
深度学习算法依赖于神经网络,这是一个在20世纪40年代首次提出的计算模型,在其中,类似神经元的节点层模仿了人类大脑如何分析信息。费城宾夕法尼亚大学计算生物学家Casey Greene表示,直到大约五年前,基于神经网络的机器学习算法还需要研究人员将原始信息处理为更有意义的形式,然后再将其输入到计算模型中。但是,来自智能手机快照或大规模基因组测序等来源的数据集数量激增,并且算法创新使人类有可能退后一步。机器学习的这一进步(“深度”部分)迫使计算机(而不是其人类程序员)找到嵌入像素和基数中的有意义的关系。随着神经网络中各层对信息的过滤和排序,它们也相互通信,从而使每一层都可以优化前一层的输出。
最终,此过程允许训练有素的算法分析新图像并将其正确识别为例如Charles Darwin或患病细胞。但是,随着研究人员远离算法,他们将无法控制分类过程,甚至无法准确解释软件的功能。尽管这些深度学习网络可以非常准确地做出预测,但Finkbeiner说:“有时候,要弄清网络所能做出的良好预测,仍然是一项挑战。”
直到现在,包括成像在内的许多生物学子学科都从这些预测中获益。十年前,用于自动生物图像分析的软件专注于测量一组图像中的单个参数。例如,在2005年,安妮·卡彭特(Anne Carpenter)马萨诸塞州剑桥市麻省理工学院和哈佛大学的计算生物学家发布了一个名为CellProfiler的开源软件包,以帮助生物学家定量测量各个特征:例如,显微镜领域中荧光细胞的数量或长度的斑马鱼。
但是深度学习使她的团队走得更远。她说:“我们一直在测量生物学家不知道自己想从图像中测量的东西。”记录并结合视觉特征(例如DNA染色,细胞器质地和细胞中空白空间的质量)可以产生成千上万的“特征”,其中任何一个都可以揭示新的见解。当前版本的CellProfiler包含一些深度学习元素,她的团队希望在明年添加更复杂的深度学习工具。
“大多数人都很难解决这个问题,”卡彭特说,“但是,在单个细胞图像中,与在细胞群体的转录组分析中一样,有很多信息,实际上可能更多。 ”
这种处理方式使Carpenter的团队采取了一种较少监督的方法,将细胞图像转化为与疾病相关的表型,并加以利用。 Carpenter是犹他州盐湖城递归制药公司的科学顾问,该公司正在使用其深度学习工具来针对罕见的单基因疾病进行药物开发。
挖掘基因数据
当涉及深度学习时,不是任何数据都可以。该方法通常需要大量的,注释良好的数据集。成像数据提供了自然拟合,基因组数据也是如此。
旧金山的Verily Life Sciences(以前称为Google Life Sciences)。谷歌母公司Alphabet的子公司Verily的研究人员和谷歌开发了一种深度学习工具,该工具可以比传统工具更准确地识别一种常见的遗传变异类型,即单核苷酸多态性。该软件称为DeepVariant,可将基因组信息转换为类似图像的表示形式,然后将其分析为图像(请参阅“深潜工具”)。谷歌基于深度学习的基因组研究负责人马克·德普里斯托斯(Mark DePristo)希望DeepVariant对于研究主流之外的生物的研究人员特别有用-那些低质量的参考基因组和高错误率的基因变异识别方法。与植物公司DeepVariant一起工作时,他的同事Ryan Poplin的错误率已接近其他方法中20%的典型值(接近2%)。
Verily Life Sciences(前身是谷歌生命科学)也一直使用此类数据。Verily是Google母公司Alphabet的子公司,研究人员开发了一种深度学习工具,可以比传统工具更准确地识别常见的遗传变异类型,即单核苷酸多态性。这种名为DeepVariant的软件,将基因组信息转换成图像表示形式,然后将其作为图像进行分析。
多伦多加拿大深度基因组学公司首席执行官Brendan Frey也致力于研究基因组数据,但其目标是预测和治疗疾病。多伦多大学的弗雷(Frey)的学术团队开发了受过健康细胞基因组和转录组数据训练的算法。这些算法建立了这些数据中RNA加工事件(例如剪接,转录和聚腺苷酸化)的预测模型。 Frey说,即使将这些算法应用于临床数据,它们也能够识别突变并将其标记为病原体,尽管他们从未见过临床数据。在Deep Genomics,Frey的团队正在使用相同的工具来识别和确定软件发现的疾病机制,以开发源自短核酸序列的疗法。
药物开发是另一项具有适合深度学习的海量数据集的学科。在这里,深度学习算法正在帮助解决分类难题,筛选诸如形状和氢键之类的分子特征,从而确定对这些潜在药物进行排名的标准。例如,总部位于旧金山的生物技术公司Atomwise开发了将分子转换为3D像素网格(称为体素)的算法。这种表示方式使该公司能够以原子精度解释蛋白质和小分子的3D结构,并建模诸如碳原子的几何形状之类的特征。该公司首席执行官亚伯拉罕·海菲茨(Abraham Heifets)说,这些特征随后被转化为数学向量,该算法可用于预测哪些小分子可能与给定的蛋白质相互作用。他说:“我们所做的许多工作都是针对没有已知结合物的[蛋白质]目标。”
Atomwise正在使用这种策略来推动其新的AI驱动的分子筛选程序,该程序扫描1000万个化合物的库,为学术研究人员提供多达72种潜在的感兴趣的蛋白质小分子结合剂。
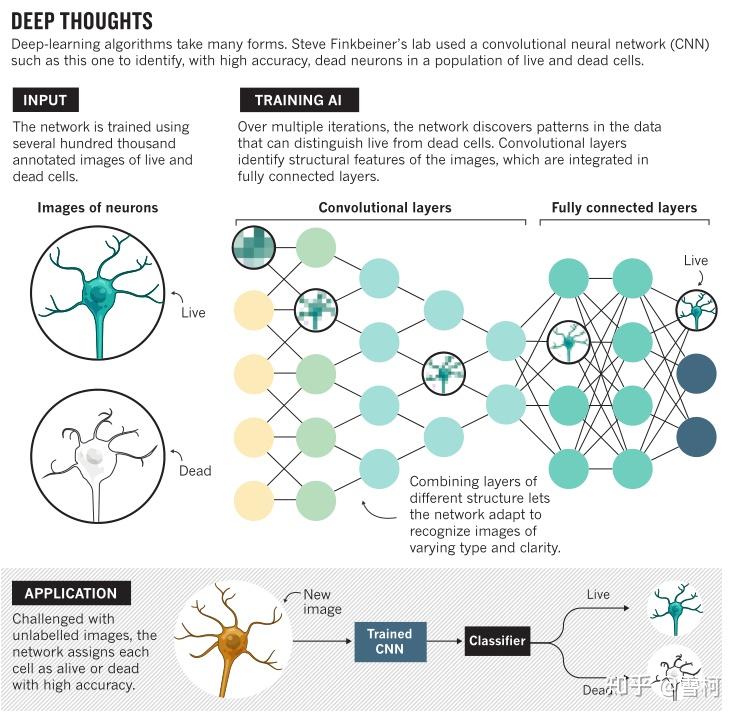
深度学习工具还可以帮助研究人员对疾病类型进行分层,了解疾病亚群,找到新的治疗方法,并将其与适合的患者进行临床测试和治疗。例如,Finkbeiner是一个名为“回答肌萎缩侧索硬化症”的组织的成员,该组织致力于整合来自1000名肌萎缩侧索硬化症(也称运动神经元疾病)患者的一系列数据——基因组学、转录组学、表观基因组学、蛋白质组学、影像学甚至多功能干细胞生物学。他说:“我们将第一次拥有一个数据集,可以应用深度学习,看看深度学习能否揭示我们在培养皿中测量的细胞周围的东西与病人身上发生的事情之间的关系。”
挑战和警告
研究人员警告称,尽管深度学习前景广阔,但它带来了重大挑战。与任何计算生物学技术一样,算法产生的结果只能和输入的数据一样好。模型对训练数据的过度拟合也是一个问题。此外,对于深度学习而言,数据数量和质量的标准往往比一些实验生物学家可能预期的更为严格。
深度学习算法需要非常大的数据集,这些数据集需要很好的注解,这样算法才能学会区分特征和分类模式。更大的,明确标记的数据集-有数百万代表不同实验和生理条件的数据点——为研究人员训练算法提供了最大的灵活性。Finkbeiner指出,在他的工作中,通过大约15000个例子,算法训练得到了显著改善。卡彭特说,这些高质量的“ground truth”数据可能异常难以获得。
为了避免这一挑战,研究人员一直在研究以更少的数据进行更多训练的方法。 Carpenter说,底层算法的进步使神经网络可以更有效地使用数据,从而可以对一些应用程序进行少量图像的训练。科学家还可以利用转移学习,即神经网络将从一种数据类型获取的分类能力应用到另一种类型的能力。例如,芬克拜纳(Finkbeiner)的团队开发了一种算法,该算法最初用于根据形态变化预测细胞死亡。尽管研究人员对其进行了训练以研究啮齿动物细胞的图像,但首次将其暴露于人类细胞的图像时,其准确性达到了90%,随着经验的积累,它的准确性提高到了99%。
对于Google的某些生物图像识别工作,Google Accelerated Science使用的算法最初是针对从互联网上提取的数亿张消费者图像进行训练的。然后,研究人员使用与他们希望研究的图像类似的少至数百张生物学图像来完善该训练。
谷歌加速科学的研究科学家米歇尔·戴蒙指出,计算机既不聪明也很懒惰。他们缺乏从正常变异中区分生物学相关差异的判断力。她指出:“计算机在发现批量变化方面出奇地出色。”因此,要获得将被输入深度学习算法的数据,通常意味着要对实验设计和控制施加很高的标准。谷歌加速科学要求研究人员将对照随机放置在细胞培养板上,以考虑诸如培养箱温度等微妙的环境因素,并使用生物学家可能使用的两倍数量的对照。戴蒙打趣道:“我们让移液管的移动变得很困难。"
戴蒙说,这种危险凸显了生物学家和计算机科学家合作设计包含深度学习的实验的重要性。在谷歌的最新项目中,这种细致的设计变得更加重要:等高线。等高线是一种对细胞成像数据进行聚类的策略,以突出趋势(如剂量反应),而不是将它们分类(如活的或死的)。
格林警告说,尽管深度学习算法可以在没有人类先入之见和过滤器的情况下评估数据,但这并不意味着它们是公正的。训练数据可能会被扭曲——例如,当只使用来自北欧的基因组数据时就会发生这种情况。在这些数据上训练的深度学习算法将获得嵌入的偏见,并在预测中反映出来,这反过来可能导致不平等的病人护理。如果人类帮助验证这些预测,就提供了对问题的潜在检查。但如果让一台计算机独自做出关键决策,这种担忧就会令人不安。格林说:“把这些方法看作是增强人类能力的一种方式,比把它们看作是替代人类的一种方式要好。”
接下来的挑战是理解这些算法是如何构建特征的,或者说是它们首先用来分类数据的特征。马里兰州巴尔的摩市insilo Medicine的研究科学家Polina Mamoshina说,计算机科学家正在通过改变模型中的个体特征,然后检查这些调整如何改变预测的准确性来解决这个问题。insilo Medicine利用深度学习来改进药物发现。但是不同的神经网络处理同一个问题的方式是不一样的,格林警告说。他说,研究人员越来越关注能够做出准确和可解释预测的算法,但目前系统仍是黑盒子。
格林说:“我认为高度可解释的深度学习模型不会在2018年出现,尽管我希望自己是错的。”
参考文献:Webb S . Deep learning for biology[J]. Nature, 2018, 554(7693):555-557. nature的这篇文章不会是一个终结,2019年一篇名为:生物信息学的深度学习:大数据时代的介绍、应用和展望再次打破了沉寂。
让我们一起来领略一下
摘要 深度学习在处理大数据方面尤为强大,在包括生物信息学在内的各个领域都取得了巨大成功。随着生物学大数据时代的推进,可以预见,深度学习在该领域将变得越来越重要,并将被纳入绝大多数分析管道中。在这篇综述中,我们提供了深度学习的开放性介绍,以及它在生物信息学中的典型应用的具体例子和实现。本文从生物信息学领域深度学习的最新研究成果入手,指出了适合深度学习的问题。之后,我们以一种简单易懂的方式介绍了深度学习,从浅层神经网络到传说中的卷积神经网络、传说中的递归神经网络、图神经网络、生成对抗性网络、变分自动编码器以及最新的体系结构。然后,我们提供了8个实例,涵盖了生物信息学的五个研究方向和所有四种数据类型,并用TensorFlow和Kera编写了实现。最后,我们讨论了用户在采用深度学习方法时会遇到的共性问题,如过度匹配和可解释性,并提出了相应的建议。
免费获取地址:https://github.com/lykaust15/Deep_learning_examples.
引言 随着计算能力的显著提高和大数据的进步,深度学习已经成为近年来最成功的机器学习算法之一。它一直在不断刷新许多机器学习任务的最新表现,并促进了许多学科的发展。例如,在计算机视觉领域,基于卷积神经网络的方法已经占据了图像识别,目标检测,图像inpainting和超分辨率三个主要方向。在自然语言处理领域,基于递归神经网络的方法通常代表了从文本分类到语音识别和机器翻译的广泛任务的最新性能。在高能物理、心理学等多个方面领域都有广泛的应用。
深度学习显然已经证明了在推动生物信息学领域的能力,包括序列分析,结构预测和重建,生物分子性质和功能预测,生物医学图像处理和诊断,生物分子相互作用预测和系统生物学。具体来说,关于序列分析,人们使用深度学习预测非编码序列变异的影响,模型转录因子结合亲和力景观,提高DNA测序和肽测序[分析DNA序列修改,和模型各种post-transcription监管事件,如可变聚腺苷酸化,可变剪接,转录起始站点,非编码RNA。在结构预测方面,利用深度学习预测蛋白质二级结构;采用深度学习来模拟蛋白质与其他分子相互作用时的结构;利用深度神经网络预测蛋白接触图和膜蛋白的结构;通过将深度学习与贝叶斯推断相结合,提高了荧光显微镜的超分辨率。在生物分子性质和功能预测方面,利用深度学习预测酶的Commission number (EC number)来预测酶的详细功能;利用深度学习预测蛋白质基因本体(GO);通过深度学习预测蛋白亚细胞位置。在使用深度学习来执行生物医学图像处理和生物医学诊断方面也有一些突破。例如,提出了一种基于深度神经网络的方法,在皮肤癌分类方面可以达到皮肤科医生的水平;利用迁移学习解决数据饥渴问题,促进医疗诊断自动化;提出了一种深度学习方法,可以从未标记生物样本的透射光图像中自动预测荧光标记;也提出了深度学习方法进行分析。
除了不断增长的计算能力和改进的算法之外,深度学习在生物信息学领域成功的核心原因是数据。在生物学领域产生的大量数据曾经被认为是一个巨大的挑战,实际上使得深度学习非常适合于生物学分析。特别是,深度学习在处理以下生物数据类型方面显示出了优越性。首先,深度学习已经成功地处理了序列数据,如DNA序列、RNA序列、蛋白序列和纳米孔信号。通过使用反向传播和随机梯度下降训练,深度学习在检测和识别隐藏在序列数据中的已知和之前未知的motifs、pattern和domains方面是专家。递归神经网络和带一维滤波器的卷积神经网络适合处理这类数据。然而,由于递归神经网络发现的模式不易解释和可视化,如果想要找出神经网络发现的隐藏模式,卷积神经网络通常是生物序列数据的最佳选择。其次,深度学习在处理二维和张力类数据方面尤其强大,如生物医学图像和基因表达谱。标准卷积神经网络及其变种,如残差网络、密连网络和双径网络,在处理生物医学数据方面表现出了令人印象深刻的性能。借助卷积层和池化层,这些网络可以系统地检查不同尺度下隐藏在原始映射中的模式,并将原始输入映射到一个自动确定的隐藏空间,其中的高层表示具有丰富的信息,适合于监督学习。第三,深度学习还可用于处理图形数据,如症状-疾病网络、基因共表达网络、蛋白-蛋白相互作用网络和细胞系统层,并促进了最新性能。处理网络的核心任务是进行节点嵌入,可以进行下游分析,如节点分类、交互预测、社区检测等。与浅层嵌入相比,基于深度学习的嵌入以树的方式聚合节点邻居的信息,参数较少,并且能够纳入领域知识。可以看出,上述三种数据都是原始数据,在我们将数据输入到模型时,没有经过太多的特征提取过程。深度学习非常擅长处理原始数据,因为它可以端到端的方式进行特征提取和分类,从而自动确定重要的高层特征。对于已经经过特征提取过程的结构化数据,深度学习可能不会显著提高性能。然而,只要仔细调整超参数,它不会比传统的方法(如SVM)差。
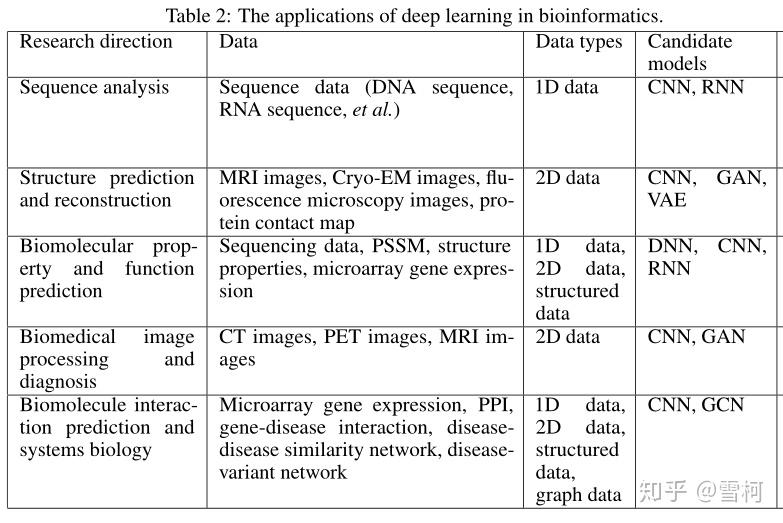
深度学习在生物信息学的应用
考虑到深度学习在促进生物信息学研究方面的巨大潜力,为了促进其发展和应用,在本综述中,我们将首先详细深入地介绍深度学习(第2节),从浅层神经网络到深层神经网络及其上述变体,适用于生物数据分析。之后, 我们提供一些具体的例子(第3节),并在Github上实现, 包括五个生物信息学研究方向(序列分析,结构预测和重建,生物分子属性和功能预测,生物医学图像处理和诊断,以及生物分子相互作用预测)和系统生物学)和所有四种类型的数据(1D序列,2D图像和轮廓,图形和预处理数据)。
在网络类型方面,这些例子将涵盖全连接神经网络、标准卷积神经网络(CNN)、递归神经网络(RNN)、残差网络(ResNet)、生成对抗网络(GAN)、变分自编码器(VAE)和图卷积神经网络(GCN)。在这些具体的例子之后, 我们将讨论研究人员在使用深度学习时可能遇到的潜在问题以及相应的可能解决方案(第4节),包括过度拟合(第4.2节),数据问题(第4.1和4.3节),可解释性(第4.4节),不确定性缩放(第4.5节),灾难性遗忘(第4.6节)和模型压缩(第4.7节)。
从浅层神经网络到深度学习 在这一节中,我们将首先介绍浅神经网络的形式及其核心组件(第2.1节)。之后,我们介绍了标准CNN和RNN的关键组成部分(2.2节)。由于标准的CNN和RNN在过去几年中得到了极大的改进,我们还将介绍几种最先进的架构(第2.3节),包括ResNet、DenseNet和SENet。在介绍了规则的一维和二维数据架构之后,我们介绍了用于处理网络数据的图神经网络(第2.4节)。然后,我们介绍了两种重要的生成模型(2.5节),GAN和VAE,它们可以用于生物医学图像处理和药物设计。最后,我们概述了当前可用的框架,这些框架使得构建深度学习模型非常方便(第2.6节)。
图1是浅层神经网络的结构。
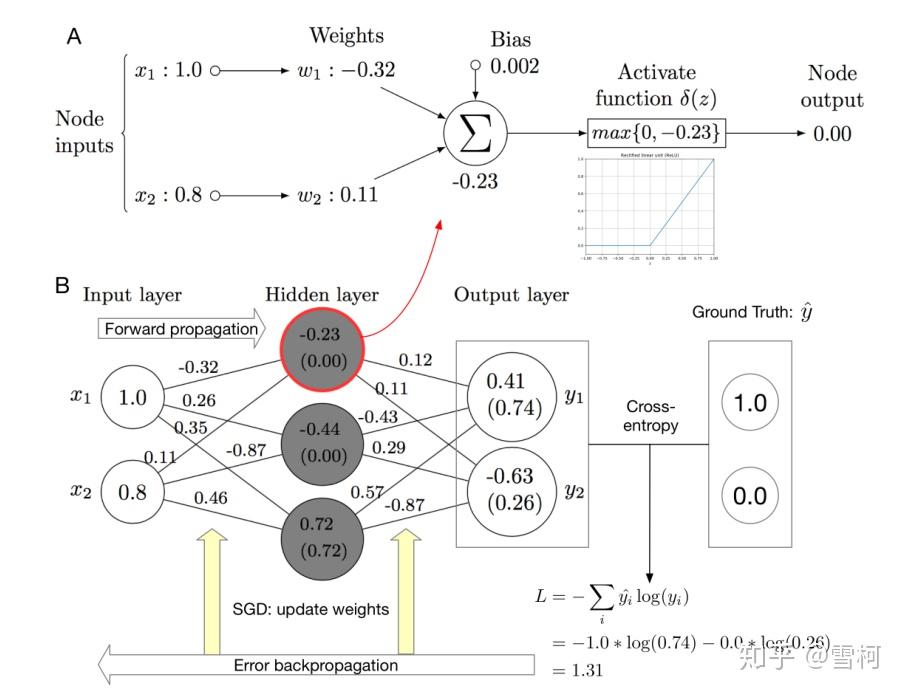
图2 详细地展示了CNN内部的卷积层和其结构。
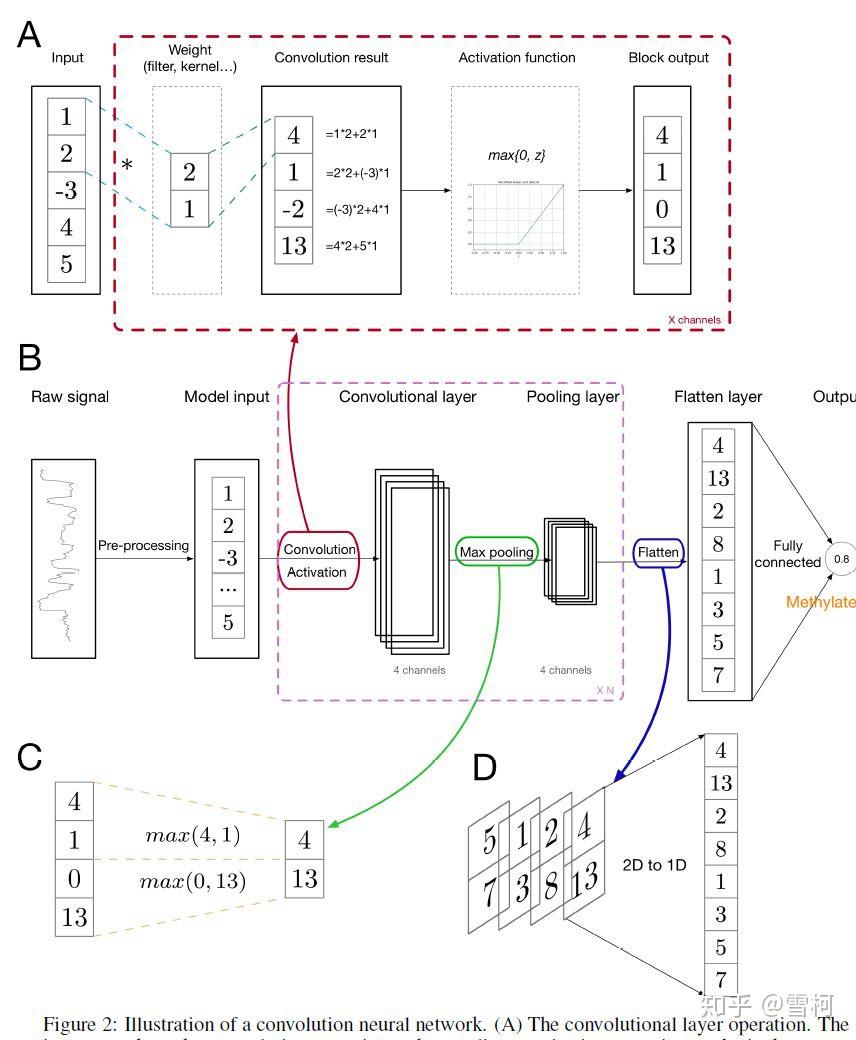
图3展示了循环神经网络结构。
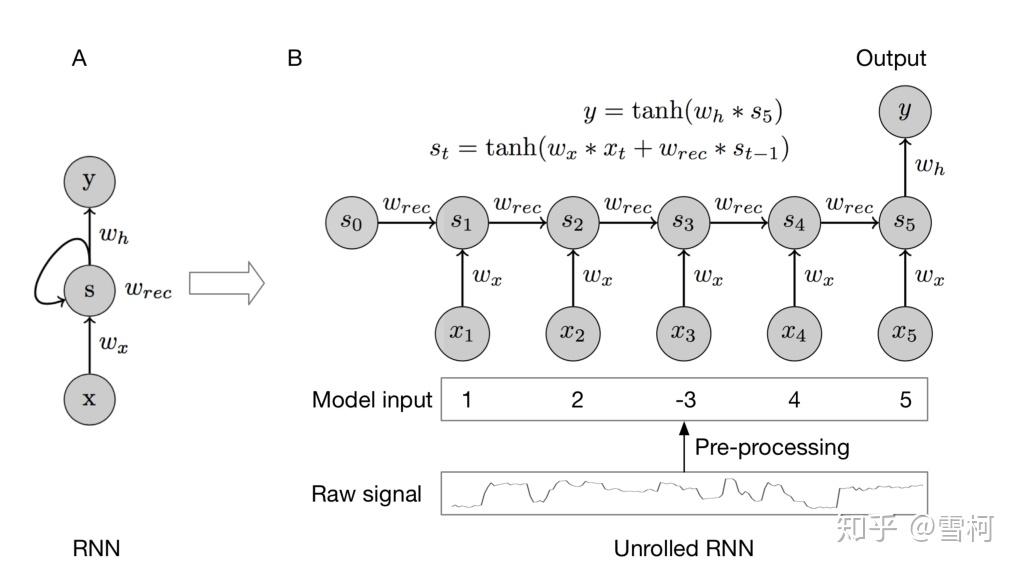
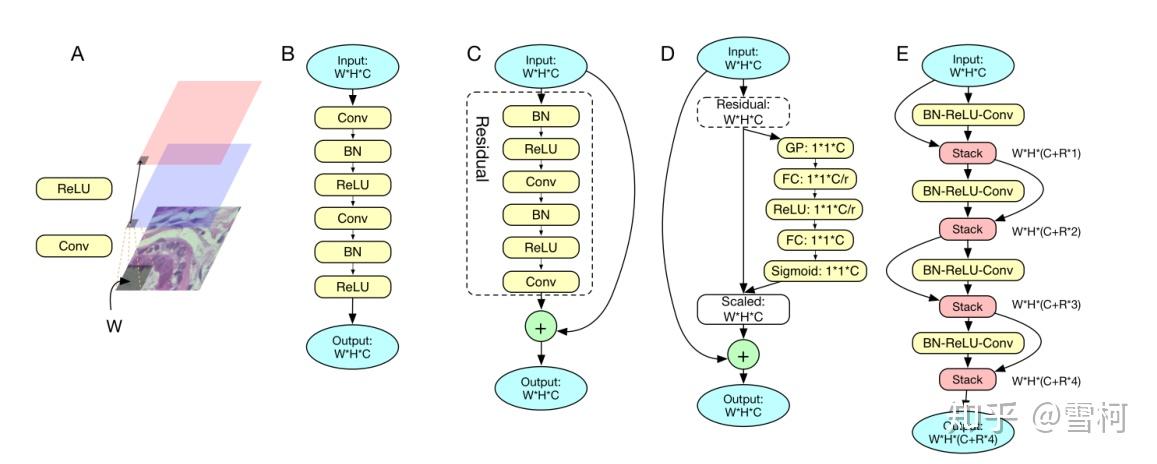
图4展示了前沿的不同卷积神经网络在图像应用上的深度网络架构。
在2.4节中,本文简要介绍用图神经网络来处理网络数据,网络数据是生物信息学中常见的数据类型。
图5展示了图神经网络的应用示例。(A)图形数据的典型示例。(B)嵌入空间。在该嵌入空间中,每个数据点由向量表示,而(A)中的原始拓扑信息保留在该向量中。(C)用于在(A)中嵌入网络的图神经网络。我们使用节点a和b作为示例。 每个节点的内部属性被视为原始表示。 在每一层中,节点聚合来自其邻居的信息,并使用平均和激活函数更新表示。
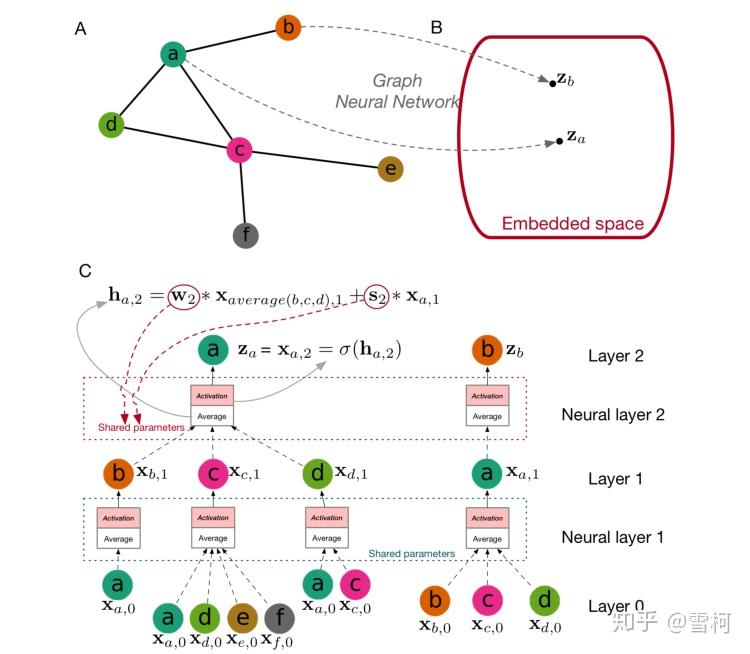
图6是GAN的图示。在GAN中,我们有一对网络同时相互竞争。生成器网络负责生成新的数据点(本例中的酶序列)。判别器网络试图将生成的数据点与实际数据点区分开。
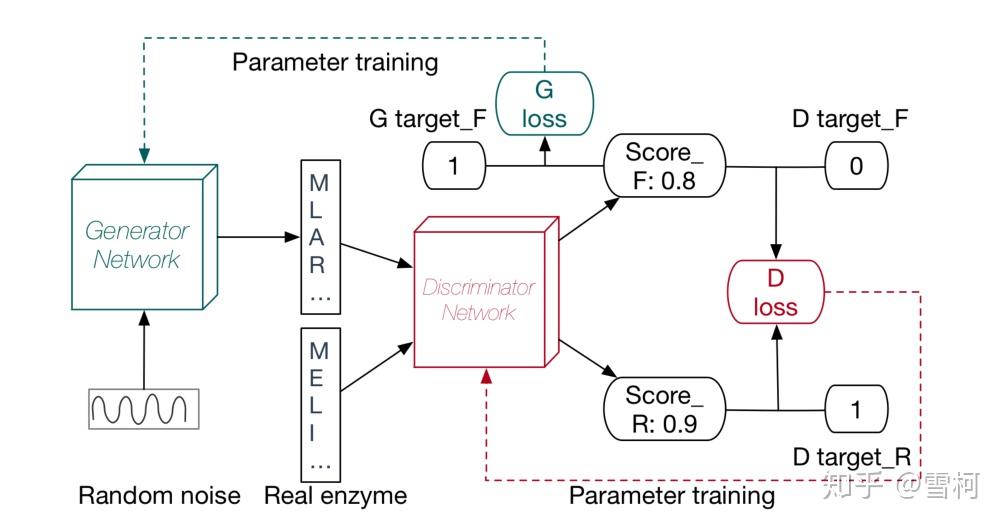
图7:(A)自编码器示意图,由编码器和解码器组成。编码器网络将输入压缩到潜在向量中,解码器网络从潜在向量中重构数据。(B)变分自编码器说明。
代码实现 地址:https://github.com/lykaust15/Deep_learning_examples
深度学习是一种非常强大和有用的技术,它促进了包括生物信息学在内的各个领域的发展。随着生物大数据时代的推进,为了进一步推动深度学习在生物信息学中的应用,在这篇综述中,我们首先回顾了深度学习的成果。之后,我们从浅神经网络,到传奇的卷积神经网络,传奇的递归神经网络,图神经网络,生成对抗神经网络,变分自编码器,做了简单而易懂的介绍。我们还提供了详细的示例和实现,以方便研究人员采用和开发基于深度学习的自己的方法。最后,我们指出了使用深度学习的常见困难并提出了相应的建议。虽然本综述没有涵盖深度学习的所有方面,如深度强化学习和深度学习的理论方面,但它涵盖了生物信息学中深度学习应用的大部分方面。我们相信,这篇综述将有助于阐明深度学习在生物信息学、生物医学和医疗保健领域的未来发展和应用。
参考文献 [1] Li Y , Huang C , Ding L , et al. Deep learning in bioinformatics: Introduction, application, and perspective in the big data era[J]. Methods, 2019.
部分内容引用:
【30页综述和代码实现】 基于深度学习的生物信息学应用及展望 作者:专知 来源:微信 深度学习的使用会更大的解放生物学家的双手,也建议大家有所了解,这两篇文献在大多数平台都可以免费下载,是生信入门深度学习的一份宝贵的资料。目前深度学习并不能解决所有的问题,面对不同的问题解决得效果也有很大得差别,期待一些新的突破。
你希望下一个还是AlphaX吗?评论区谈一谈你的看法,国内哪个团队或者公司可能会在这一领域取得更大得突破。 |
|
 /3
/3 